專任:張文綺教授

最後更新日期 :
2026-02-24
| 姓名 | 張文綺 |
|---|---|
| 職稱 | 教授 |
| 電話 | +886-6-2757575 ext.58311 |
| sarah321@mail.ncku.edu.tw | |
| 研究專長 | 生物資訊、基因體學、總基因體學 |
| PBMB Lab | http://wcchang.itps.ncku.edu.tw/ |
 學歷
學歷
2003 – 2008 博士.國立清華大學生物資訊與結構生物研究所
2001 – 2008 碩士.國立清華大學生命科學系
 經歷
經歷
- 2018.08 – present
教授.國立成功大學熱帶植物與微生物科學研究所 - 2013.02 – 2017.07
副教授.國立成功大學熱帶植物科學研究所 - 2009.08 – 2013.01
助理教授.國立成功大學熱帶植物科學研究所 - 2008.08 – 2009.07
博士後研究.國立交通大學生物資訊及系統生物研究所
 實驗室簡介
實驗室簡介
我們利用資訊方法處理生物的大數據,建立植物基因調控網路與生物資訊分析系統。亦將生物資訊方法應用於許多植物科學的研究上。
 研究方向
研究方向
1. 生物資訊資料庫與軟體開發
2. 生物資訊應用研究與生物大數據分析
- 基因轉錄調控機制探討與調控網路之建立 (我們著重在promoter、transcription factor以及histone modification對基因表達操控之研究)
- 非編碼RNA(non-coding RNA)對基因表達調控之探討
- 土壤微生物與植物交互作用之研究
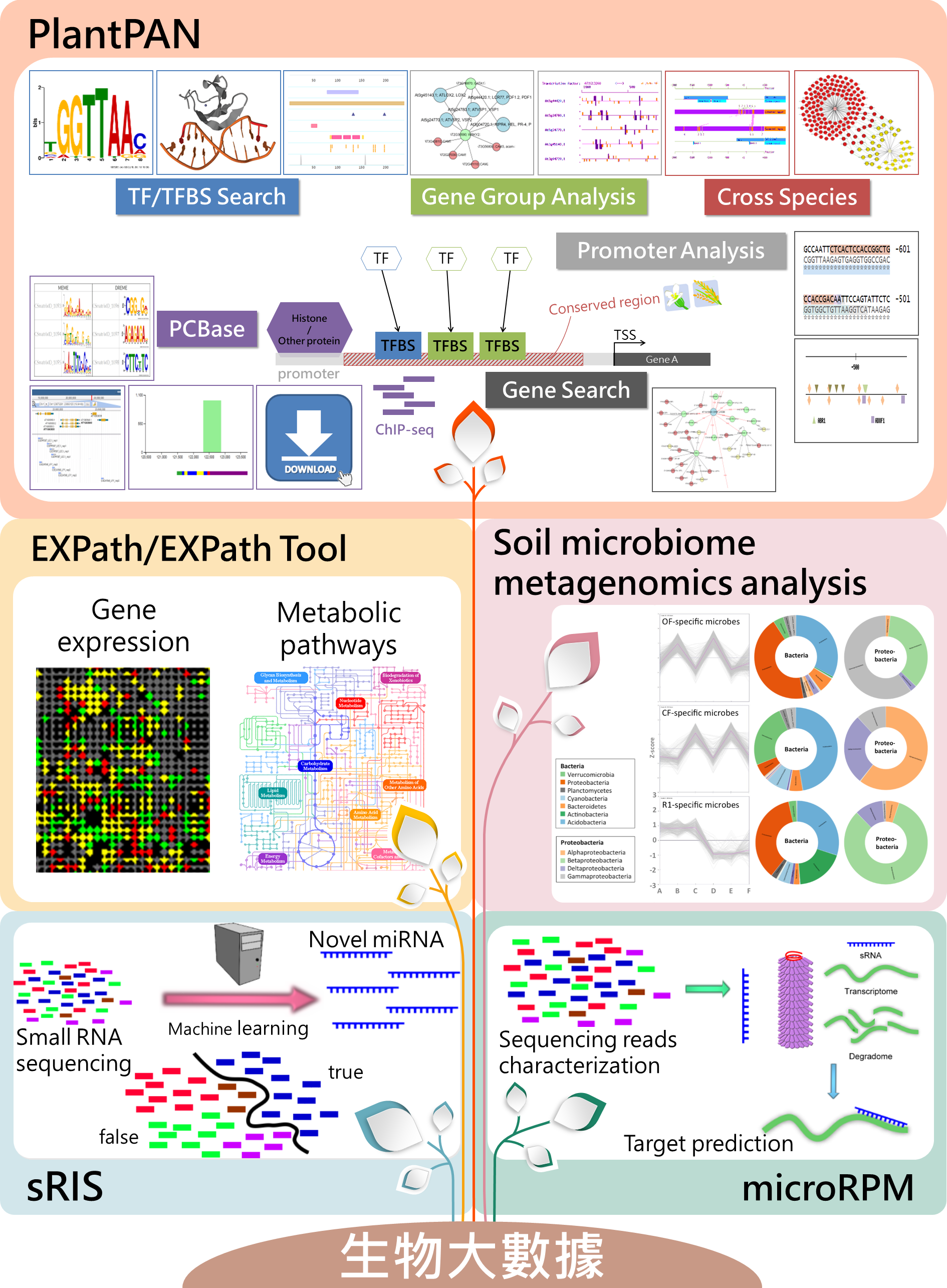
- PlantPAN:具備最完整之植物TF-TFBS資料庫,可分析基因轉錄調控網路與提供調控因子資訊,並進行跨物種的分析比較。
- EXPath/EXPath Tool:快速從高通量基因表現資料(如:生物晶片、次世代定序)中辨別差異表達基因並分析基因功能以及其所參與之代謝途徑。
- Soil microbiome metagenomic analysis:與農改場合作利用次世代定序技術探討長期有機與慣行農法對作物之產量、抗病性以及土壤永續性之影響。
- sRIS:利用轉錄組(transcriptome)、sRNA定序資料、降解組(degradome)定序資料及病毒的基因組資料了解植物基因、微小RNA與病毒RNA (vsiRNA)之間的調控關係。
- microRPM:利用機器學習方法所開發之微小RNA預測系統。可直接透過小RNA(sRNA)定序資料預測已知與未知的微小RNA (microRNA)。
 榮譽獎項
榮譽獎項
- 榮獲國立成功大學112學年度通識課程優良教師獎
- 2017 榮獲楊祥發院士傑出農業科學年輕學者獎
- 2018 榮獲成功大學教學優良教師獎
- 2018 指導學生榮獲農業資訊科技最佳碩士論文獎
- 近五年發表SCI期刊論文共14篇,5-year-IF點數共88.87,每篇平均6.35,其中 3篇IF > 10,Ranking<5%
 代表著作
代表著作
- Chow et al., "PlantPAN 3.0: a new and updated resource for reconstructing transcriptional regulatory networks from ChIP-seq experiments in plants". Nucleic Acids Research, 47(D1):D1155-1163, 2019. (IF: 10.162, Rank: 4.90% in BIOCHEMISTRY & MOLECULAR BIOLOGY) (以通訊作者身分發表)
- Hou et al., "Paddy-upland rotation for sustainable agriculture with regards to diverse soil microbial community", Scientific Reports, 8(1):7966, 2018. (以通訊作者身分發表)
- Tseng et al., "microRPM: A microRNA Prediction Model based only on plant small RNA sequencing data", Bioinformatics, 34(7), 1108-1115, 2018. (IF:7.307, Rank: 3.51% in MATHEMATICAL & COMPUTATIONAL BIOLOGY) (以通訊作者身分發表)